Coordinateurs : Joe Miller (Global Biodiversity Information Facility), Copenhagen Denmark et Vanessa Terra, Universidade Federal de Uberlândia - UFU
Introduction
Open Tree of Life a pour objectif de construire un arbre de la vie complet, dynamique et disponible dans un format digital, en synthétisant les arbres phylogénétiques publiés ainsi que des données taxonomiques. Ce projet peut-être considéré comme la version phylogénétique de GBIF qui est une entité combinant les données provenant de milliers de jeux de données de biodiversité dans un système permettant à tout un chacun d’accéder à ces données, que ce soit avec un intérêt géographique ou taxonomique donné. OToL utilise les données en accès ouvert de la même manière, puisque nous n’avons pas d’arbre phylogénétique incluant toutes les formes de vies. Les utilisateurs de OToL peuvent télécharger une sous-partie de l’arbre, par exemple un arbre de toutes les Légumineuses (Fabaceae) ou d’une liste d’espèces. Tout comme pour GBIF, plus les données entrantes sont de bonne qualité, plus celles sortantes le seront. Les arbres OToL peuvent être utilisés pour de multiples utilisations, incluant la recherche et la visualisation.
Il est important de comprendre que l’arbre synthétique de OToL inclus des branches provenants d’analyses phylogénétiques intégrant de nombreuses sources, et comprend ainsi différents gènes, morphologies et échantillonnage. De plus, plusieurs branches dans l’arbre sont uniquement connues grâce à la taxonomie, ce qui signifie qu’elles ne sont, à l’heure actuelle, représentés dans aucune phylogénie source dans OToL. OToL utilise un service de résolution des noms taxonomiques robuste (Taxonomic Name Resolution Service (TNRS)) afin de placer ces branches dans l’arbre. Par exemple, le clade des Fabaceae (OTT: 560323) contient 24 479 espèces mais seulement 4835 espèces sont placées grâce à des phylogénies. Les espèces qui ne sont dans aucun arbre source sont placées dans une polytomie au niveau du genre. Afin de résoudre ces ploytomies, nous avons besoin d’inclure autant d’arbres de haute qualité que possible. Il est également possible via OToL de télécharger un arbre incluant uniquement les branches issues d’analyses phylogénétiques. Pour en apprende davantages sur OToL, vous pouvez consulter la section À Propos du site de Open Tree of Life.
Emily Jane McTavish (UC Merced and an OToL PI) nous a fourni de l’information, incluant les arbres synthétiques des Légumineuses et les listes ci-dessous. Notre objectif est de travailler avec Emily Jane McTavish afin d’améliorer le clade des Légumineuses dans OToL. Puisque il est aisé d’importer des phylogénies depuis Treebase dans OToL, la récupération des analyses sur les Légumineuses dans Treebase est le premier endroit où travailler afin d’améliorer l’arbre synthétique de OToL.
Statut des Légumineuses dans OToL
| Phylogénie dérivée de ToL (version Dec 2019) | # points terminaux | lien vers le fichier |
|---|---|---|
| Toutes les espèces de Fabaceae | 24,479 | OToL |
| Seulement basé sur les phylogénies | 4,835 | Newick |
-
Analyses publiées présentement intégrées dans la version OTol actuelle
-
Analyses partiellenement révisées dans OToL et contenant des espèces de Fabaceae
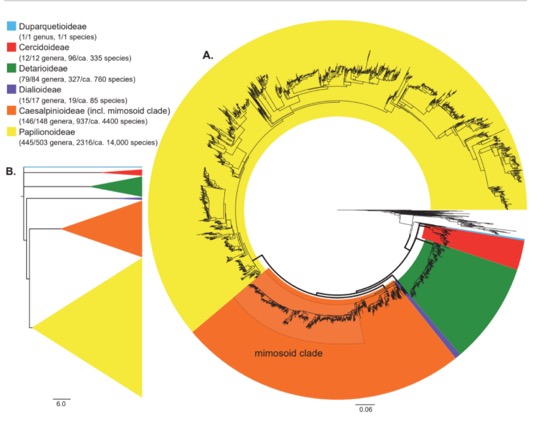
En prenant en considération les arbres dans OToL et la distribution des personnes intéressées dans le projet, il serait opportun de commencer à travailler sur ces sous-familles : Caesalpinioideae (incl. le clade des mimosoid), Dialioideae, Detarioideae, Cercidoideae et Duparquetioideae.
Voici ce dont nous disposions en Janvier 2021 :
Ceci est un tableur Google listant le statut des arbres dans OToL et quelques arbres cibles potentiels à impoter :
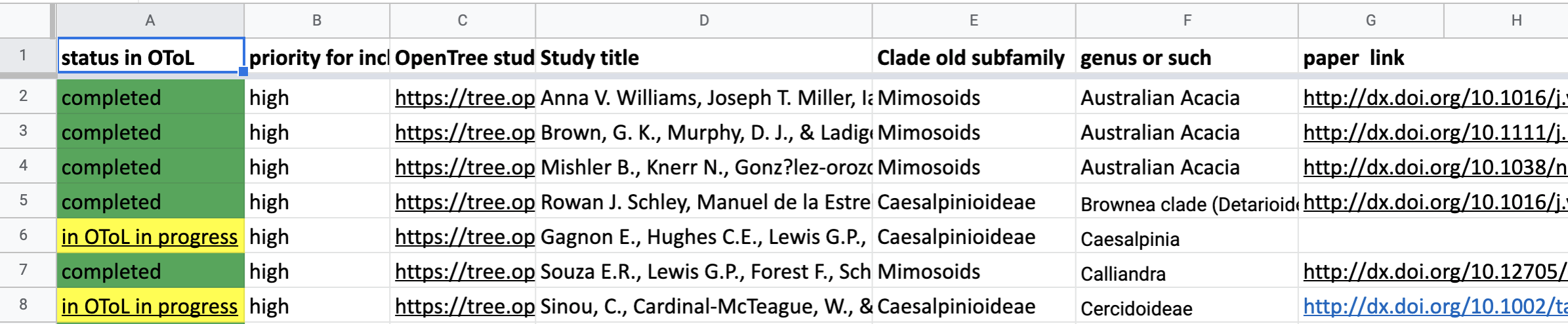
| Tab | Statut | Action |
|---|---|---|
| Liste OToL | Completé | Déjà dans OToL, aucune action nécessaire |
| Liste OToL | En cours | Considérant nos connaissances sur le clade et la littérature, est-ce vraiment à inclure ? |
| Liste OToL | Priorité | Mettre à jour la priorité dans la colonne 2 |
| Arbres Potentiels | Dans Treebase | Importer dans OToL depuis TreeBase et révision |
| Arbres Potentiels | Pdf, pas dans treebase | Determiner si prioritaire, trouver arbres ou contacts ou y a-t-il de nouvelles études sur ce clade ? |
| Arbres Potentiels | À évaluer | Cela en vaut-il la peine ? Y a-t-il de nouvelles études sur ce clade ? |
| Arbres Potentiels | Pdf à trouver | Cela en vaut-il la peine ? Y a-t-il de nouvelles études sur ce clade ? |
| Suggérer un arbre | Besoin d’arbres | Récents arbres couvrant ce groupe pas dans l’arbre synthétique actuel, surtout au niveau des espèces |
/* Arbres potentiels : Cette liste d’arbres a été obtenue principalement de citations de LPWG. C’est une liste ancienne qui a besoin d’être mise à jour.
Liste des priorités pour le Groupe de Travail
- Identifier les études devant être incluses de manière prioritaire
- niveau spécifique
- absent de l’arbre synthétique actuel
- publications récentes
- arbres permettant de résoudre le squelette taxonomique, nouvelles analyses phylogénomiques.
- dans Treebase ou facilité à récupérer des fichiers d’arbres
- Identifier les arbres de la liste OToL et la liste des Arbres Potentiels qui valent la peine d’être inclus
- Télécharger le fichier de l’arbre actuel de OToL en Newick et trouver les lacunes dans la couverture
- Mettre à jour la taxonomie dans le tableur
- afin d’aider à trouver les lacunes
- afin d’éliminer les erreurs
- Mettre l’emphase sur les Caesalpinioideae (incl. le clade des Mimosoids), Dialioideae, Detarioideae, Cercidoideae et Duparquetioideae
- afin d’obtenir une meilleure couverture dans ces clades
Nous avons besoin de votre aide !
Comment réviser les fichiers dans OToL
Cliquer sur le lien d’étude d’OpenTree afin de vous familiariser avec l’interface. Vous aurez besoin d’un compte GitHub afin de faire des modifications. De plus amples détails seront ajoutés plus tard. L’arbre synthétique de OToL n’est mis à jour qu’occasionnellement. La mise à jour la plus récente date de mai 2024.
Idées de publications
Nous pourrions réécrire un article décrivant l’avant et l’après de ce projet. Combien d’espèces et d’études avons-nous ajoutées ? Dans quelle mesure la phylogénie OToL est-elle meilleure/utile ? Nous pourrions fournir des exemples de la manière dont l’arbre pourrait être utilisé, en particulier dans le cadre des groupes de travail sur la taxonomie et les occurrences. Le GBIF travaille sur un visualiseur phylogénétique qui a déjà été ajouté au portail de données sur les légumineuses.
Rejoignez le Groupe de Travail
Veuillez envoyer un courriel à Joe Miller ou Vanessa Terra ou tout simplement commencez à regarder pour des analyses à inclure. Aucune expérience dans la révision des fichiers OToL n’est nécessaire. La chose la plus importante que vous pouvez nous fournir est votre connaissance des meilleurs arbres à inclure.